|
|
MULTIMEDIA FILES / ARCHIVOS MULTIMEDIA
|
2024.
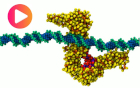
STAG2-RAD21 Complex: a Unidirectional DNA Ratchet Mechanism in Loop Extrusion (1/2).
STAG2-RAD21-DNA Complex: Leftward full-turn displacement. Full 3 microsecond trajectory of the coarse-grained STAG2-RAD21-DNA biased simulation with a 8.75 kcal mol-1 Angstrom-1 force constant, with the target set to the left. When the biasing potential is applied to the left, the protein complex moved along the DNA molecule. The guide region slipped from an initial position in contact with the minor groove of the DNA helix to the next minor groove, while the pawl region moved equivalently from its initial position in contact with the major groove of the DNA molecule to the next major groove.
Complejo STAG2-RAD21-ADN: Desplazamiento de vuelta completa de ADN hacia la izquierda. Trayectoria completa de 3 microsegundos de la simulación de grano grueso de STAG2-RAD21-DNA impulsada con una constante de fuerza de 8,75 kcal mol-1 Angstrom-1, con la posición final fijada a la izquierda. Cuando el potencial de empuje se aplica hacia la izquierda, el complejo proteico se desplaza a lo largo de la molécula de ADN. La región guía se desliza desde una posición inicial en contacto con el surco menor de la hélice de ADN hasta el siguiente surco menor, mientras que la región trinquete se mueve de forma equivalente desde su posición inicial en contacto con el surco mayor de la molécula de ADN hasta el siguiente surco mayor.
Published in: / Publicado en: International Journal of Biological Macromolecules 276, 133822. [PubMed] [https://doi.org/10.1016/j.ijbiomac.2024.133822]
|

|
2024.
STAG2-RAD21 Complex: a Unidirectional DNA Ratchet Mechanism in Loop Extrusion (2/2).
STAG2-RAD21-DNA Complex: Rightward full-turn displacement. Full 3 microsecond trajectory of the coarse-grained STAG2-RAD21-DNA biased simulation with a 8.75 kcal mol-1 Angstrom-1 force constant, with the target set to the right. When force is exerted to the right, the situation is the opposite: the biasing potential is able to partially displace the guide region from the minor groove of the DNA helix to an intermediate position between the adjacent major and minor grooves. The pawl region, however, remains engaged to the same major groove and is unable to move to the next major groove. As a result, rightward displacement does not occur, and the protein remains stuck in a closed, clamp-like conformation.
Complejo STAG2-RAD21-ADN: Desplazamiento de vuelta completa de ADN hacia la derecha. Trayectoria completa de 3 microsegundos de la simulación de grano grueso de STAG2-RAD21-DNA impulsada con una constante de fuerza de 8,75 kcal mol-1 Angstrom-1, con la posición final fijada a la derecha. Cuando la fuerza se ejerce hacia la derecha, la situación es la contraria: el potencial de empuje es capaz de desplazar parcialmente la región guía desde el surco menor de la hélice de ADN hasta una posición intermedia entre los surcos mayor y menor adyacentes. La región de trinquete, sin embargo, queda fijada en el mismo surco mayor y es incapaz de moverse al siguiente surco mayor. Como resultado, no se produce el desplazamiento hacia la derecha y la proteína permanece anclada en una conformación cerrada tipo pinza.
Published in: / Publicado en: International Journal of Biological Macromolecules 276, 133822. [PubMed] [https://doi.org/10.1016/j.ijbiomac.2024.133822]
|
|
2017.
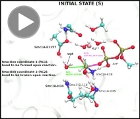
Two-step ATP-driven opening of cohesin head (1/3).
ATP hydrolysis at active site 1 of human cohesin in the presence of Rad21. Quantum Mechanics / Molecular Mechanics. The different steps in the reaction are
highlighted: initial structure (S), stabilization of the catalytic water molecule, transition state (TS) and final product (P).
Hidrólisis de ATP en el sitio activo 1 de cohesina humana en presencia de Rad21. Mecánica Cuántica / Mecánica Molecular. Se
indican los diferentes pasos de la reacción: estructura inicial (S), estabilización de la molécula de agua catalítica, estado de
transición (TS) y producto final (P).
Published in: / Publicado en:
Scientific Reports 7, 3266. [PubMed] [https://doi.org/10.1038/s41598-017-03118-9]
YouTube: [https://youtu.be/ZsPWPVCUiG8] |
|
2017.
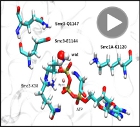
Two-step ATP-driven opening of cohesin head (2/3).
Positioning of Smc1A-K1120. Molecular Dynamics. After ATP hydrolysis at active site 1 of human cohesin, aminoacid Smc1A-K1120 moves close to the catalytic water molecule in
the active site 2 and remained in its new location in a stable conformation.
Recolocación de Smc1A-K1120. Dinámica Molecular. Tras la hidrólisis de ATP en el sitio activo 1 de cohesina humana, el aminoácido Smc1A-K1120
se acerca a la molécula de agua catalítica del sitio activo 2 y permanece en su nueva localización en una conformación estable.
Published in: / Publicado en:
Scientific Reports 7, 3266. [PubMed] [https://doi.org/10.1038/s41598-017-03118-9]
YouTube: [https://youtu.be/edfNbtQK8ZA] |
|
2017.
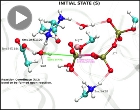
Two-step ATP-driven opening of cohesin head (3/3).
ATP hydrolysis at active site 2 of human cohesin. Quantum Mechanics / Molecular Mechanics. The different steps in the reaction are
highlighted: initial structure (S), transition state (TS) and final product (P).
Hidrólisis de ATP en el sitio activo 2 de cohesina humana. Mecánica Cuántica / Mecánica Molecular. Se
indican los diferentes pasos de la reacción: estructura inicial (S), estado de transición (TS) y producto final (P).
Published in: / Publicado en:
Scientific Reports 7, 3266. [PubMed] [https://doi.org/10.1038/s41598-017-03118-9]
YouTube: [https://youtu.be/hVyNMAhOxMo] |
|
2012.
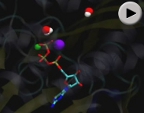
Molecular Dynamics simulation of GTPase interfaces of FtsZ tetramer. .
Short filament of bacterial protein FtsZ: Behaviour of GTP (sticks), Mg2+, (green sphere), K+ (purple sphere) and water molecules
(red and white structures) at the three interfaces of a FtsZ tetramer over 8 nanoseconds of unrestricted molecular dynamics.
Filamento corto de la proteína bacteriana FtsZ: Comportamiento de las moléculas de GTP (bastones), Mg2+, (esfera verde), K+ (esfera púrpura) y agua
(estructuras en rojo y blanco) en las tres interfaces de un tetrámero de FtsZ durante 8 nanosegundos de dinámica molecular no restringida.
Published in: / Publicado en:
FEBS Letters 586, 1236–1239. [PubMed] [https://doi.org/10.1016/j.febslet.2012.03.042]
YouTube: [https://youtu.be/AC7d7zRP314] |
|
2011.
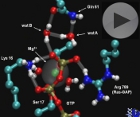
The role of Gln61 in ras p21 GTP hydrolysis: a QM/MM study. .
Quantum Mechanics / Molecular Mechanics interface: video demonstrating proton movements from water molecule A to water molecule B and then to GTP in the active centre of the protein of human oncogene Ras.
Interfaz Mecánica Cuántica / Mecánica Molecular: video mostrando el salto de un protón de la molécula de agua A a la molécula de agua B y de ahí al GTP en el
centro activo de la proteína del oncogén Ras humano.
Published in: / Publicado en:
Biophysical Journal 102, 152-157. [PubMed] [https://doi.org/10.1016/j.bpj.2011.11.4005]
YouTube: [https://youtu.be/TvmYAZtMo4c] |
|
2013.
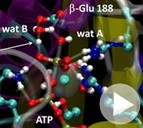
Simulation of catalytic water activation in mitochondrial F1-ATPase using a hybrid quantum mechanics/molecular mechanics
approach: An alternative role for ß-Glu 188. .
Quantum Mechanics / Molecular Mechanics interface: video demonstrating proton movements from water molecule A to water molecule B and then to ATP in the active centre of mitochondrial F1-ATPase.
Interfaz Mecánica Cuántica / Mecánica Molecular: video mostrando el salto de un protón de la molécula de agua A a la molécula de agua B y de ahí al ATP en el
centro activo de la proteína F1-ATPasa mitocondrial.
Published in: / Publicado en:
Biochemistry 52, 959-966. [PubMed] [https://doi.org/10.1021/bi301109x]
YouTube: [https://youtu.be/Ks5hZLf6ENY] |
BIOWEB: MOLECULAR MODELING GROUP
Centro de Biología Molecular Severo Ochoa
(CBM, CSIC-UAM)
http://bioweb.cbm.uam.es |
C/ Nicolás Cabrera, 1. Campus UAM, Cantoblanco
28049 Madrid. Spain
Tel: (+34) 91 196 4662 / 4663
Fax: (+34) 91 196 4420
|
|